近日,我校计算机科学与技术学院袁晓辉教授团队与广州大学孔凡江教授、刘宝辉教授团队,中科院遗传发育所田志喜研究员团队及澳大利亚塔斯马尼亚大学James Weller教授团队协作,以武汉理工大学为通讯单位在国际遗传学顶级期刊《Nature Genetics》发表了题为“Stepwise selection on homeologous PRR genes controlling flowering and maturity during soybean domestication”的最新研究成果。
该研究不仅进一步完善了长日照条件下大豆光周期的分子调控网络,阐明了大豆适应高纬度生态环境的遗传基础;而且发现了大豆驯化过程中同源基因的逐步进化与选择的分子机制,从而确认了光周期开花是作物核心的驯化性状。
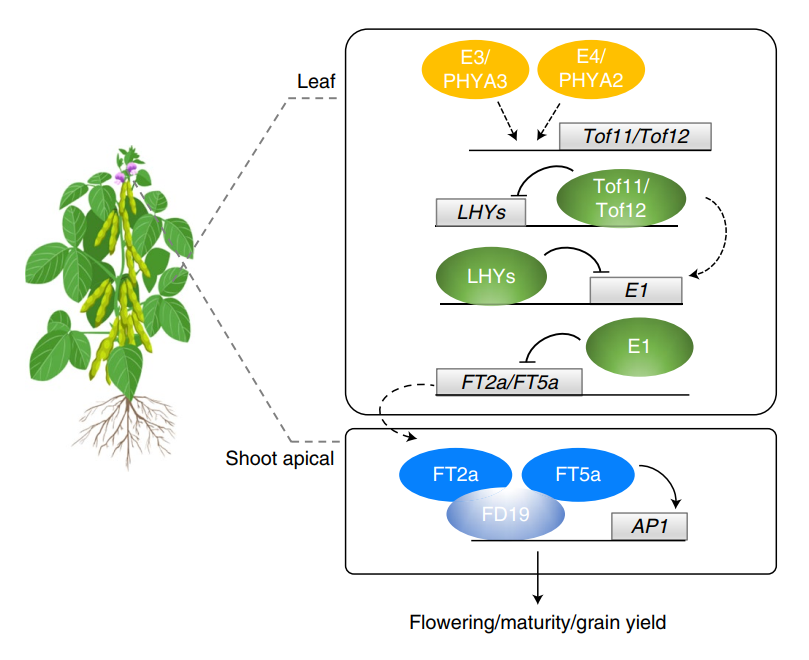
图1 大豆光周期开花和产量形成的分子模式图
该研究利用大数据基因组学分析、生物信息学和经典正向遗传学相结合的方法,发掘了两个长日照条件下控制开花期的关键位点Tof11和Tof12。分子机制解析表明,Tof11和Tof12通过调控LHY和E1基因控制大豆光周期开花,建立了完整的光周期调控分子网络(图1)。研究同时发现,Tof11和Tof12发生了渐进式的变异和人工选择。其中,Tof12-1的功能缺失突变首先被强烈选择,使栽培品种的开花期和成熟期普遍提前;Tof11-1的功能缺失型突变的发生于Tof12-1之后,在Tof12-1遗传背景上再次受到选择,从而进一步缩短了栽培大豆的开花期和生育期,因此提高了栽培大豆的适应性和种植。该研究首次系统报道了作物驯化过程中开花期基因的进化与选择分子机制。
《Nature genetics》中文名为《自然·遗传学》, 该杂志主要发表高质量的遗传学研究。包括对人类和植物性状以及其他模式生物的遗传和功能基因组研究,最新影响因子为25.455。
我校计算机科学与技术学院2015年自引进袁晓辉教授成立“环境与生物信息大数据团队”以来,打破传统从北海道大学、香港城市大学、康奈尔大学、德国马普、中科院等国内外著名高校引进计算机、生物、农业等学科的青年才俊,形成一支特色鲜明的交叉学科团队。近年来,团队成员在Nature Genetics、Genome Biology、TKDE、AAAI等生物和计算机学科顶级期刊发表论文30余篇,参与和主持国家重点研发计划、国家自然科学基金、“神农”大科学装置等国家重点项目10余项。
参考文献:
Lu, S., Dong, L., Fang, C. et al. Stepwise selection on homeologous PRR genes controlling flowering and maturity during soybean domestication. Nat Genet (2020). https://doi.org/10.1038/s41588-020-0604-7